一个70公斤的成人一共有37万亿个细胞,每一个细胞都不完全相似【1】。这些多样化的细胞承载着我们所有的生命活动,但研究起来却并不容易。一是人体组织材料有限,二是难以体外培养原态细胞。三是难以获得特异类型的细胞。直到1951年,Hela细胞系的建立,才真正开启了对人类细胞活动分子层面上的科学研究。但是,如何研究体内状态下人体细胞分子水平上的特征和功能,仍然是一个棘手的问题。进十年来,高通量DNA分子测序结合生物统计分析成为了研究这一问题的有效手段。DNA分子测序技术可以检测在DNA和染色质层面上发生的遗传变异和转录翻译调控。利用DNA测序产生的大量数据,结合数据来源的细胞特质或人类性状进行相关性分析,是研究人体组织细胞各种表型的分子机制的重要手段。
人体组织器官的很多功能都需要多种细胞同时参与协作完成。单细胞技术能够直接对所有组织中的细胞无偏差地研究,并为统计分析提供大量样本数据。随着测序技术的灵敏性增加,单细胞DNA测序和单细胞RNA测序都大大推动了单细胞水平上的研究,帮助人们重新在细胞种类丰富的系统中(如神经系统【2】,免疫系统【3】)鉴定到全新的细胞类型。同时,也促进了研究材料稀缺的领域(如胚胎【4】,生殖细胞【5】,癌症循环细胞【6】)的进展。在肿瘤领域,单细胞技术不仅揭示了癌症内部的细胞发生过程【7】,还鉴定到了癌症干细胞的存在以及肿瘤微环境的差异【8】。但是怎么在单细胞水平上,研究每个细胞的主要转录因子和调控元件,以及它们对应的靶点基因呢?
2018年8月2日,来自华盛顿大学的Jay Shendure(HHMI研究员)组和Cole Trapnell组合作在Cell上发表了题为A Single-Cell Atlas of In Vivo Mammalian Chromatin Accessibility的论文,在哺乳动物模式生物小鼠的多个组织上,为我们展示了如何通过单细胞的染色质调控信息来研究转录调控。

单细胞测序技术(single cell sequencing,SCS)的发展历史【9】
文章通过Jay Shendure组2015年开发的split-pool-split单细胞ATAC-seq技术(ATAC-seq能检测基因组上转录因子结合后形成的开放位点来鉴定增强子和该细胞重要的转录因子),检测了小鼠13种组织中85种特异的全基因组染色质状态。目前高通量的单细胞ATAC-seq技术主要分两种,一是在96孔板中通过split-pool-split的方法标记单细胞【10】,可以适用于大部分实验室但是数据质量还有待提高,另一种是通过实验仪器形成单个液体来包裹单细胞进行标记【11】,依赖仪器但是数据质量相对较好。总的来说,单细胞数据信号噪音较高,分析需要大量统计学手段,这篇文章的一个亮点就是探索了很多新的数据分析方法,来试图解析单细胞中转录调控的问题。
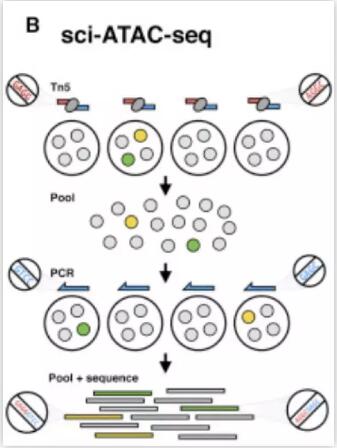
单细胞ATAC-seq的原理:通过两次标记来避免两个细胞带上同样的标签。
分析单细胞ATAC-seq数据的一个难点是怎么确定细胞的种类,文章结合此前发表的小鼠单细胞RNA-seq数据【12】,尝试了基因种类分析,基因相关性打分和转移标签等几种不同的分析方法来探索这个问题。并发现这些方法都各有优劣。值得一提的是,一些实验室也在积极探索在同一个细胞中检测RNA和chromatin信息,相关工作发表在预印本杂志bioRxiv上【13】。其次,文章详细鉴定了各个细胞类别主要的转录因子以及各个增强子对应的靶点基因,为未来研究各个细胞的发育调控奠定了基础。另外,文章发现了一些有趣的现象,比如同样是来源心脏的表皮细胞,其染色质就有三种不同状态,而且可以推测其中两种分别是受Flt4和Notch通路的调控,这说明它们在心脏中可能具有不同功能。最后,作者发现在小鼠单细胞数据当中鉴定到的染色质开放区域较大量细胞鉴定到的开放染色质区域能够更富集人类GWAS位点,说明单细胞在类别细分后,鉴定到的增强子可能具有更强的细胞功能特异性。
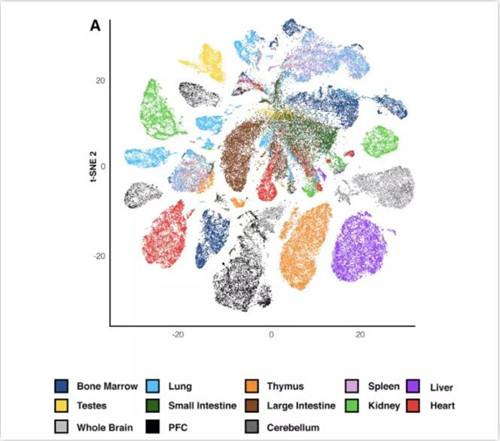
tSNE能够通过染色质信息将大部分不同组织来源的细胞区分开来
这篇文章最后提到了数据的信噪比,种类分析和实验方法上的一些局限性,为未来的改进提供了方向。之前,国内外的部分实验室已经开始就将单细胞开放染色质定位技术运用到不同的组织器官上,来研究胚胎发育【14,15】和神经系统发育【16,17】中的转录调控问题,我们也期待在这些研究中鉴定到新的转录因子和调控元件的功能,能够得到进一步实验的验证。另外,在基因组和转录水平上的研究,还仅仅是解释细胞多样性的冰山一角,怎么研究人体不同组织器官中各个细胞类型的蛋白质种类和丰度,蛋白质修饰和蛋白相互作用的差异,还亟待技术的突破。我们期待新的单细胞技术,结合传统生物学手段,帮助我们最终解析人类细胞多样性的形成和功能。

论文的两位共同通讯作者
专家点评
颉伟(清华大学生命科学学院研究员,清华-北大生命科学联合中心PI,HHMI国际学者,国家“杰青”)
每个人体有几十万亿的细胞,而每个细胞在功能、形态等方面都是不同的。如何研究和阐明每个细胞的功能是生物学的基本核心问题之一。单细胞RNA-seq的迅速发展大大增加了我们对已有细胞的认识以及未知新的细胞类型的发现(国内像汤富酬、郭国骥等实验室在这方面做出了杰出的贡献)。然而RNA-seq信息中通常会缺失的一个重要信息是基因组调控序列。包括增强子、启动子、绝缘子等调控序列是促成组织和发育时空特异性基因表达的重要调节因子。基于染色质的一系列表观遗传调控方法的进步为研究这些问题提供了很多新的工具。其中ATAC-seq是近年来比较常用的一个检测染色质开放性的方法,这种技术是斯坦福大学的William Greenleaf和Howard Chang实验室于2013年开发的。通常调控序列的染色质会处于比较开放的状态,很适合比较广谱的研究顺式调控序列和基因表达调控。
华盛顿大学的Jay Shendure实验室前期开发了大量的单细胞研究工具,包括single cell ATAC-seq。在这个工作就是他们通过single cell ATAC-seq来研究小鼠多种组织单细胞水平的染色质状态和基因调控的研究做出的一个尝试。在技术上,作者采用index组合的方法,进一步提高单细胞ATAC-seq的通量。通过该方法,作者描绘了13个小鼠组织中大约100,000个单细胞的开放染色质图谱,并且成功区分了诸多组织中的大部分细胞类群,也为相关组织的研究提供了很好的参考和借鉴。另外,作者通过单细胞ATAC-seq的数据,鉴定了许多组织特异性的转录因子。最后,基于人与鼠的开放染色质的保守性,作者揭示了部分人类遗传病与开放染色质间的潜在关系,拓展了该研究的意义。
一个有趣的问题是能否将大量不同组织的单细胞RNA-seq和单细胞ATAC-seq整合起来进行比较研究。由于这些数据是不同的单细胞实验获得的,而每个细胞的identity在实验中都是推测出来的,而并非完全明确的,因此在技术上整合两种数据具有很大的挑战性。作者采用了一种label-transfer的方法,整合RNA-seq和ATAC-seq数据对细胞进行聚类,并将每个single cell ATAC-seq中的细胞周围的single cell RNA-seq细胞的最常见的label赋予该细胞。值得一提的是在分析中用到的single cell RNA-seq数据之一就是郭国骥实验室今年在Cell 发表的小鼠组织single cell RNA-seq的数据。这种label-transfer的分析方法具有探索性,从结果来看有合理性的部分,同时也有明显的问题,比如还缺乏其他独立实验结果的验证等。
总之,在该项研究中,作者通过sci-ATAC-seq以及相应分析和质控方法,获得了较好的多组织单细胞层面的开放染色质信息,为组织结构,发育和分化,各组织器官的调控网络的研究提供了丰富的参考。然而,目前相关技术仍然有一些缺陷,比如相比single cell RNA-seq,single cell ATAC-seq每个细胞的coverage比较低。另外,每种或每类细胞的identity如何验证?是否有方法能够有效的在单细胞水平同时检测转录组和染色质动态调控。最后,受限于物种差异,以小鼠开放染色质的数据直接分析部分人类遗传性疾病可能带有一定局限性。相信这项研究是探索基因如何在单细胞水平实现时空表达特异性调控这一激动人心的核心生物学问题的刚刚开始。
参考文献
1. Cusanovich, D. A. et al. A Single-Cell Atlas of In Vivo Mammalian Chromatin Accessibility Article A Single-Cell Atlas of In Vivo Mammalian Chromatin Accessibility. Cell 174, 1–16 (2018).
2. Pollen, A. A. et al. Low-coverage single-cell mRNA sequencing reveals cellular heterogeneity and activated signaling pathways in developing cerebral cortex. Nat. Biotechnol. 32, 1053–1058 (2014).
3. Jaitin, D. A. et al. Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types. Science 343, 776–9 (2014).
4. Xue, Z. et al. Genetic programs in human and mouse early embryos revealed by single-cell RNA sequencing. Nature 500, 593–7 (2013).
5. Guo, F. et al. The Transcriptome and DNA Methylome Landscapes of Human Primordial Germ Cells.Cell 161, 1437–1452 (2015).
6. Ramsköld, D. et al. Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nat. Biotechnol. 30, 777–782 (2012).
7. Tirosh, I. et al. Single-cell RNA-seq supports a developmental hierarchy in human oligodendroglioma. Nature 539, 309–313 (2016).
8. Venteicher, A. S. et al. Decoupling genetics, lineages, and microenvironment in IDH-mutant gliomas by single-cell RNA-seq. Science (80-. ). 355, (2017).
9. Wang, Y. & Navin, N. E. Advances and applications of single-cell sequencing technologies. Mol. Cell 58, 598–609 (2015).
10. Cusanovich, D. a et al. Epigenetics. Multiplex single-cell profiling of chromatin accessibility by combinatorial cellular indexing. Science 348, 910–4 (2015).
11. Buenrostro, J. D. et al. Single-cell chromatin accessibility reveals principles of regulatory variation.Nature 523, 486–490 (2015).
12. Han, X. et al. Mapping the Mouse Cell Atlas by Microwell-Seq. Cell 172, 1091–1107.e17 (2018).
13. Liu, L. et al. Deconvolution of single-cell multi-omics layers reveals regulatory heterogeneity.bioRxiv 316208 (2018). doi:10.1101/316208
14. Cusanovich, D. A. et al. The cis-regulatory dynamics of embryonic development at single-cell resolution. Nature 555, 538–542 (2018).
15. Li, L. et al. Single-cell multi-omics sequencing of human early embryos. Nat. Cell Biol. 20, 847–858 (2018).
16. Preissl, S. et al. Single-nucleus analysis of accessible chromatin in developing mouse forebrain reveals cell-type-specific transcriptional regulation. Nat. Neurosci. 21, 432–439 (2018).
17. Lake, B. B. et al. Integrative single-cell analysis of transcriptional and epigenetic states in the human adult brain. Nat. Biotechnol. 36, 70–80 (2018).