人类的致病遗传变异既有点突变又有碱基插入缺失突变【1】。2016年,哈佛大学David Liu实验室开发出新型单碱基编辑器CBE(Cytosine base editors),实现了C·G--T·A碱基对的自由转换【2】,2017年底他们又构建出ABE(Adenine Base Editor),可实现A·T--G·C碱基对的转换,这对众多点突变遗传病的治疗有重要意义,因而在基因治疗领域的应用前景相当广阔【3】(详见:突破丨Nature长文发表基因编辑最新成果——无需切割DNA也能自由替换ATGC)。不过,除了C·G--T·A和A·T--G·C碱基转换,对其它类型的碱基突变以及碱基的插入缺失突变,目前依然缺乏有效的研究工具:传统的同源重组修复(HDR)需要外源的双链/单链DNA模板,系统复杂且效率低下,这极大地限制了相关工作的开展,因此开发更加高效且广谱的精准基因编辑工具迫在眉睫。
2019年10月21日,哈佛大学David Liu实验室在Nature杂志上发表了题为Search-and-replace genome editing without double-strand breaks or donor DNA的论文。文章开发出了全新的精准基因编辑工具PE (Prime Editors),新工具PE无需额外的DNA模板便可有效实现所有12种单碱基的自由转换,而且还能有效实现多碱基的精准插入与删除(最多可插入44bp的碱基,可删除80bp的碱基),这一全能性的工具为基因编辑领域带来了重大变革。

新工具PE是以CRISPR-Cas9系统为基础,在两方面加以改造:首先是改造单链引导RNA(sgRNA),其3’末端增加了一段RNA序列,新获得的RNA被称作pegRNA;第二则是将Cas9切口酶(H840A突变型,只切断含PAM的靶点DNA链)与逆转录酶融合获得新的融合蛋白。pegRNA的3’端序列有双重角色,一段序列作为引物结合位点(PBS),与断裂的靶DNA链3’末端互补以起始逆转录过程,另一端序列则是逆转录的模板(RT模板),其上携带有目标点突变或插入缺失突变以实现精准的基因编辑(图1)。
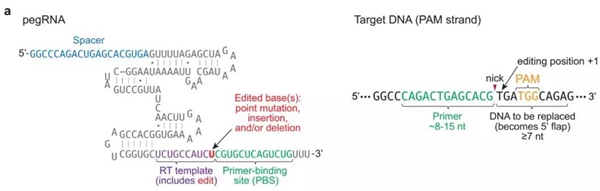
图1 改造后的pegRNA结构
基因编辑工具PE的基本原理如图2所示,首先是在pegRNA的引导下,Cas9 H840切口酶切断含PAM的靶点DNA链,断裂的靶DNA链与pegRNA的3’末端PBS序列互补并结合,之后逆转录酶发挥功能,沿RT模板序列开始逆转录反应。反应结束后DNA链的切口处会形成处在动态平衡中的5’-和3’-flap结构,其中3’flap结构的DNA链携带有目标突变,而5’flap结构的DNA链则无任何突变。细胞内5’flap结构易被结构特异性内切酶识别并切除,之后经DNA连接和修复后靶位点处便实现了精准的基因编辑。
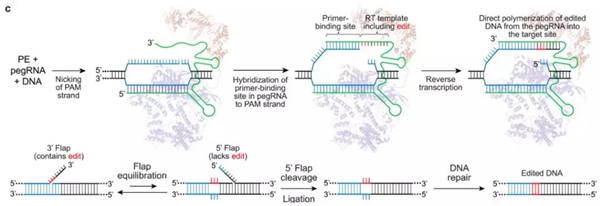
图2 基因编辑工具PE的基本原理
在经体外验证和酵母中的验证之后,研究者将野生型的鼠白血病病毒(M-MLV)逆转录酶融合在Cas9 H840切口酶的C末端,构建出了第一代精准基因编辑工具PE1,在293T细胞中PE1的点突变效率为0.7~5.5%, 碱基的增加/删除效率则为4~17%,依然有更大的提升空间。之后研究者通过优化M-MLV逆转录酶得到第二代编辑工具PE2,其点突变效率和碱基的增删效率较PE1有两倍以上的提高。

图3 PE3和PE3b的原理图:PE3增加的sgRNA识别位点是未编辑的基因组DNA,PE3b增加的sgRNA只识别编辑后的基因组DNA。
PE1/2系统只编辑双链DNA的一条链,另一条非编辑链需进一步的DNA修复以完成精准编辑。传统上,通过Cas9切口酶切断非编辑链可以有效提高该链的修复效率。为此研究者在PE2的基础上,增加可切断非编辑链的sgRNA,最终获得新的PE3和PE3b系统(图3)。新系统的编辑效率较PE2提升了近3倍,在293T细胞中的最高编辑效率可达78%。当然,由于使用了两条sgRNA,PE3的随机插入缺失(Indels)风险也随之提高,这是PE3未来需要加以改进的不足之处。
研究者对不同的工具进行比较后发现,与单碱基编辑工具CBE、ABE相比,PE3/3b的单碱基编辑在效率上略有不如,但能实现更精准的编辑;而与传统的HDR相比,PE3/3b有着更高的编辑效率和更低的Indels风险。此外,PE3/3b系统在U2OS、K562、HeLa三种细胞系以及小鼠皮层原代神经元中均能发挥精准编辑效果,这表明新系统有着广泛的适用性。
总体而言,本研究开发的新工具PE是精准基因编辑领域的重大突破,在单碱基随意转换和小片段多碱基的增删方面潜力巨大,这将极大的推动生物医学的基础研究和临床基因治疗研究。
原文链接:
https://www.nature.com/articles/s41586-019-1711-4
参考文献
1. Rees, H.A. & Liu, D.R. Base editing: precision chemistry on the genome and transcriptome of living cells.Nat Rev Genet(2018).
2. Komor AC, Kim YB, Packer MS, Zuris JA, Liu DR. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420-424 (2016).
3. Gaudelli, N.M. et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage.Nature551, 464-471 (2017).
